Ghostwriting-Service Dr. Rainer Hastedt
Einführung in die Statistik-Software R Commander
1. Ausgabe vom 5. September 2016
Daten mit dem R Commander einlesen
Regression (Ordinary Least Squares)
Grafiken speichern und anpassen
Eine Grafik in der gewünschten Größe speichern
Grafikparameter anpassen (Base Graphics)
Arbeitsschritte und Ergebnisse speichern
R-Skripte erzeugen und wiederverwenden
Der R Commander ist eine grafische Benutzeroberfläche für R - ausgereift, weitgehend selbsterklärend und ebenso wie R kostenlos. Zielgruppe sind Nutzer, die nur gelegentlich mit Statistik-Software arbeiten und ein intuitiv zu bedienendes System bevorzugen, vergleichbar mit SPSS.
Mein Tutorial soll zum selbständigen Arbeiten mit dem R Commander befähigen. Hierzu bespreche ich alle meiner Ansicht nach für die Beherrschung des R Commanders bedeutsamen Arbeitstechniken, die Sie anhand meiner Beispiele sehr leicht an Ihrem Computer nachvollziehen können. Ich bespreche auch fortgeschrittene Techniken, sofern sie für gelegentliche Nutzer interessant sind, insbesondere zur Anpassung von Grafiken.
Mein Tutorial behandelt nur einen kleinen Teil der über den R Commander zugänglichen statistischen Methoden. Trotzdem: Wenn Sie sich mit meinem Tutorial beschäftigt haben, dann sollte es leicht für Sie sein, den R Commander auch für andere Aufgaben zu nutzen, zum Beispiel für eine Cluster-, Faktoren- oder Hauptkomponentenanalyse.
Installation und Setup
Meine Ausführungen sind auf Windows-Betriebssysteme abgestimmt. Hinweise für Nutzer von Mac- und Linux-Betriebssystemen finden sich in den R Commander Installation Notes.
Downloaden Sie R. Besuchen Sie hierzu die Website www.r-project.org und wählen Sie dort den Menüpunkt CRAN (Comprehensive R Archive Network). Wählen Sie einen Mirror, dann »Download R for Windows« und dort die Standardversion (»base«). Speichern Sie die Installationsdatei (.exe) auf Ihrer Festplatte.
Starten Sie die Installation durch einen Doppelklick auf die heruntergeladene Datei. Akzeptieren Sie grundsätzlich alle von R vorgeschlagenen Einstellungen. Weichen Sie hiervon nur ab, wenn Sie sehr genau wissen was Sie tun.
Im Dialogfeld »Komponenten auswählen« haben Sie folgende Optionen:
- Core Files
- 32-bit Files
- 64-bit Files
- Message translations
Die Core Files sind erforderlich. Wenn Sie die Optionen 32-bit Files und 64-bit Files markieren, installieren Sie R doppelt, einmal in der 32-Bit-Version und einmal in der 64-Bit-Version. Die 32-Bit-Version ist meines Erachtens nur sinnvoll, wenn Sie ein veraltetes Betriebssystem haben, für das 64-Bit-Software ungeeignet wäre. Für Windows 8 oder 10 halte ich die 64-Bit-Version für angemessen. Auf die Message translations würde ich verzichten, um immer den genauen Wortlaut der Messages zu erhalten.
Sie werden gefragt, ob Sie die Startoptionen anpassen möchten. Antworten Sie bitte mit ja. Sie erreichen dann das Dialogfeld Anzeigeoptionen.
Zur Auswahl stehen:
- MDI (ein großes Fenster) = multiple-document interface
- SDI (mehrere Fenster) = single-document interface
Wählen Sie die für den R Commander empfohlene Einstellung SDI. Sie erreichen dadurch, dass das R-Commander-Fenster in der Regel über der R-Konsole liegt.
Starten Sie R, sobald Sie die Installation abgeschlossen haben. Gehen Sie zum Menüpunkt Packages / Install package(s)…, wählen Sie einen Mirror und dann das Package Rcmdr (R Commander). Installieren Sie Rcmdr und alles, was Ihnen hierbei vorgeschlagen wird.
Starten Sie den R Commander manuell. Wählen Sie hierfür den Menüpunkt Packages / Load package … und dort das Package Rcmdr. Es kann sein, dass der R Commander beim erstmaligen Start weitere Software vorschlägt oder auf das Fehlen von Software hinweist. Installieren Sie diese Software gegebenenfalls.
Sobald Sie hiermit fertig sind:
Beenden Sie R und damit auch den R Commander. Antworten Sie auf die Frage, ob Sie ein Workspace Image speichern wollen mit ja oder nein. (Ich werde auf diese Frage weiter unten eingehen.)
Sie können den R Commander durch kostenlose Packages erweitern. Alle derartigen Packages haben einen Namen, der mit der Zeichenfolge RcmdrPlugin. beginnt. Ein solches Plugin kann im R Commander auch Optionen aus Menüs streichen oder verändern. Außerdem können zwei Plugins miteinander in Konflikt stehen, was nicht immer sofort auffallen muss.
Ich halte es für sinnvoll, zum Auftakt zwei Plugins zu installieren:
- RcmdrPlugin.EZR
- RcmdrPlugin.KMggplot2
EZR (Easy R) ersetzt die Menüs des R Commanders durch neue Menüs und verschiebt die ursprünglichen Menüs in den Menüpunkt »Original menu«, wodurch die Funktionalität der Grundversion des R Commanders erhalten bleibt.
EZR ist meines Erachtens benutzerfreundlicher als die Grundversion des R Commanders. Außerdem erweitert EZR die Funktionalität des R Commanders, zum Beispiel durch zusätzliche Grafiktypen und erweiterte Optionen für multiple Mittelwertvergleiche.
KMggplot2 ergänzt die Benutzeroberfläche des R Commanders durch das Menü KMggplot2, mit dem Sie auf einfache Weise sehr schöne Grafiken erstellen können. Wegen der Vorschaufunktion und der umfangreichen Theming-Möglichkeiten können Sie hier sehr gut experimentieren.
Starten Sie R. Wählen Sie den Menüpunkt Packages / Install package(s)… und dort RcmdrPlugin.EZR sowie RcmdrPlugin.KMggplot2 (mit gedrückter Strg-Taste können Sie mehrere Items markieren). Installieren Sie die beiden Packages.
Starten Sie anschließend den R Commander manuell (Menüpunkt Packages / Load package). Wählen Sie im R Commander den Menüpunkt Extras / Lade Rcmdr Plugin(s) … und dort die beiden Packages. Damit die Einstellung wirksam wird, müssen Sie dem vorgeschlagenen Neustart des R Commanders zustimmen.
Gehen Sie im R Commander zum Menüpunkt Extras / Speichere Rcmdr Optionen … Sie sehen jetzt ein Textfeld mit folgendem Inhalt:
###! Rcmdr Options Begin !###
options(Rcmdr = structure(list(plugins =
c("RcmdrPlugin.EZR", "RcmdrPlugin.KMggplot2" )),
.Names = "plugins") )
# Uncomment the following 4 lines (remove the #s)
# to start the R Commander automatically
# when R starts:
# local({
# old <- getOption('defaultPackages')
# options(defaultPackages = c(old, 'Rcmdr'))
# })
###! Rcmdr Options End !###
Klicken Sie auf den OK-Button. Speichern Sie die Datei unter dem vorgeschlagenen Namen im vorgeschlagenen Verzeichnis. Die Datei heißt .Rprofile und gehört in den von R als Arbeitsverzeichnis verwendeten Ordner. Beenden Sie R und damit auch den R Commander.
Öffnen Sie die Datei .Rprofile mit einem Texteditor.
R prüft beim Start, ob sich im Arbeitsverzeichnis eine Datei mit dem Namen .Rprofile befindet. Ist dies der Fall, so versucht R die in dieser Datei enthaltenen Anweisungen auszuführen.
Die erste Anweisung in .Rprofile bedeutet, dass R bei einem Start des R Commanders immer auch die Plugins EZR und KMggplot2 einbeziehen soll.
Die zweite Anweisung in .Rprofile ist wegen der #-Symbole unwirksam. Sie besagt, dass R das Package Rcmdr zu den Packages zählen soll, die beim Start immer geladen werden müssen.
Löschen Sie die #-Symbole und die Kommentare.
Die Datei .Rprofile sieht dann folgendermaßen aus:
options(Rcmdr = structure(list(plugins =
c("RcmdrPlugin.EZR", "RcmdrPlugin.KMggplot2" )),
.Names = "plugins") )
local({
old <- getOption('defaultPackages')
options(defaultPackages = c(old, 'Rcmdr'))
})
Speichern Sie die Änderungen und starten Sie R. Der R Commander müsste jetzt automatisch hochfahren, zusammen mit den beiden Plugins.
Der beschriebene Ansatz funktioniert auf meinem Computer mit EZR und KMggplot2. Ich habe auch andere Plugins ausprobiert, unter anderem das Package RcmdrPlugin.NMBU (mit linearer und quadratischer Diskriminanzanalyse). R hat auf meinem Computer mit diesem Plugin bei den Rcmdr-Options (erste Anweisung) mit einer Fehlermeldung reagiert und die Ausführung von .Rprofile abgebrochen.
Sinnvoll ist daher ein Plan B.
Sie können den Inhalt der Datei .Rprofile zur Not löschen und durch folgende Zeilen ersetzen:
.First <- function(){
require(Rcmdr)
require(RcmdrPlugin.EZR)
require(RcmdrPlugin.KMggplot2)
}
Der Name .First ist von R vorgegeben. Die Funktion .First() enthält alle Anweisungen, die R beim Start ausführen soll. R soll beim Start den R Commander laden und danach die beiden Plugins. Dies geht mit allen funktionsfähigen Plugins. Der Nachteil dieses Ansatzes (die Packages sukzessive laden) besteht darin, dass der Start des R Commanders merkwürdig aussieht - das R-Commander-Fenster öffnet sich, wird sofort geschlossen und dann wieder geöffnet.
Der Nachteil lässt sich manchmal durch eine Kombination der beiden Ansätze vermeiden. Für den R Commander mit den Plugins KMggplot2 und NMBU hat auf meinem Computer folgender Code funktioniert:
options(Rcmdr = structure( list(plugins =
"RcmdrPlugin.KMggplot2"), .Names = "plugins") )
.First <- function(){
require(RcmdrPlugin.NMBU)
}
R soll demnach den R Commander immer mit dem Plugin KMggplot2 laden. Beim Programmstart soll R das Package RcmdrPlugin.NMBU starten, was nur geht, wenn R auch das Package Rcmdr lädt.
Ich komme jetzt zu der Frage, die R Ihnen beim Beenden des Programms stellt: Wollen Sie ein Workspace Image speichern? Meiner Ansicht nach sollten Sie hierauf grundsätzlich mit nein antworten.
Bejahen Sie die Frage, so erstellt R im Arbeitsverzeichnis die Dateien .RData und .Rhistory oder ergänzt diese Dateien um zusätzliche Informationen.
- .RData enthält die von R erzeugten Objekte, die als Workspace bezeichnet werden. Zu den Objekten gehören zum Beispiel eingelesene Excel-Tabellen und lineare Regressionsmodelle
- .Rhistory enthält die von R gespeicherten Befehle, wozu auch Befehle gehören, die Sie über die grafische Benutzeroberfläche des R Commander eingeben
R lädt beim Start alle in der Datei .RData gespeicherten Objekte in den Arbeitsspeicher. Normalerweise erzeugen Sie in der aktuellen Session weitere Objekte, die R ebenfalls in den Arbeitsspeicher lädt und ebenfalls in der Datei .RData speichert, sofern Sie dies veranlassen. Sie würden Ihren Arbeitsspeicher auf diese Weise sukzessive zu einer riesigen Müllhalde machen. R hat daher im Menü Misc die Optionen List objects und Remove all objects.
Den Inhalt der Datei .Rhistory können Sie anzeigen, indem Sie das folgende Kommando in die R-Konsole schreiben und anschließend die Eingabetaste drücken:
history(max.show=Inf)
Inf bedeutet hier, dass alles gezeigt werden soll. Auch .Rhistory wird von Session zu Session größer, wenn Sie beim Beenden von R immer dem Speichern eines Workspace Images zustimmen. Der Inhalt der Datei .Rhistory ist normalerweise uninteressant, weil Sie wichtige Sachen besser speichern können, zum Beispiel mit dem R Commander als Bericht im HTML-Format oder einfach per Copy & Paste.
Es macht Sinn, die Dateien .RData und .Rhistory, falls vorhanden, zu löschen.
Sie können R in der Datei .Rprofile die Anweisung geben, auf die Frage nach dem Speichern eines Workspace Images zu verzichten. Hierzu ergänzen Sie .Rprofile um folgenden Code:
q <- function(save="no", status=0, runLast=TRUE){
.Internal(quit(save, status, runLast))
}
Der Parameterwert save="no" bedeutet hier, dass Sie beim Beenden von R auf das Speichern eines Workspace Images verzichten. Ihnen bleibt unbenommen, dies manuell zu veranlassen (Menü File der R-Konsole).
Die Datei .Rprofile hat mit der Ergänzung folgenden Inhalt:
options(Rcmdr = structure(list(plugins =
c("RcmdrPlugin.EZR", "RcmdrPlugin.KMggplot2" )),
.Names = "plugins") )
local({
old <- getOption('defaultPackages')
options(defaultPackages = c(old, 'Rcmdr'))
})
q <- function(save="no", status=0, runLast=TRUE) {
.Internal(quit(save, status, runLast))
}
Ich bin bislang davon ausgegangen, dass Sie den R Commander beenden, indem Sie die R-Konsole schließen. Dies ist die einfachste und schnellste Möglichkeit.
Wenn Sie den R Commander beenden, indem Sie das R-Commander-Fenster schließen, so werden Sie vom R Commander auf alle bedeutenden Möglichkeiten hingewiesen, Ihre Arbeitsergebnisse zu speichern.
Daten mit dem R Commander einlesen
Ausgangspunkt: Sie haben R, den R Commander, EZR und KMggplot2 erfolgreich auf Ihrem Computer installiert.
Der Import von Daten läuft über den Menüpunkt Datei / Importiere Daten, über den Sie unter anderem sehr leicht Excel- und SPSS-Datensätze einlesen können. Etwas schwieriger ist der Import von CSV-Dateien und ähnlichen Arten von Textdateien, auf den ich hier eingehen will.
CSV bedeutet Comma-separated Values und damit durch Kommata getrennte Spalten. Dies gilt für Dateien mit der englischen Schreibweise, wo Dezimalstellen durch Punkte getrennt sind.
Ich verwende als Beispiel eine CSV-Datei mit Zahlen in deutscher Schreibweise, die auf meiner Website als Download bereitsteht. Wenn Sie diese Datei in einem Texteditor öffnen, dann sehen Sie Einträge der folgenden Art:
"Typ";"Wert"
"A";27,5882287316744
"C";43,311206999949
"B";21,6038143229693
Die Datei enthält Zahlen mit Kommata als Dezimaltrennzeichen und hat zwei Spalten, die in jeder Zeile durch ein Semikolon getrennt sind. Anführungszeichen dienen hier dazu, nicht-numerische Werte zu markieren. Würden Sie die CSV-Datei mit Excel (deutsche Version) öffnen und wieder speichern, so wären die Anführungszeichen verschwunden. Dies ist normalerweise egal.
Ich will die CSV-Datei mit dem R Commander in R einlesen, Link: http://www.ghostwriting-service.de/misc/tdaten.csv. Hierzu könnte ich die Datei zunächst herunterladen, bei Bedarf mit Excel bearbeiten und dann aus meinem Arbeitsverzeichnis importieren. Ich werde die Datei stattdessen direkt von meiner Website einlesen.
Gehen Sie im R Commander zum Menüpunkt Datei / Importiere Daten / Einlesen der Textdaten aus Datei, Zwischenablage oder URL:
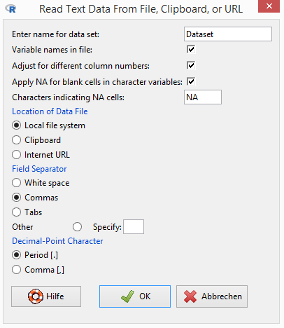
Schreiben Sie in das Textfeld »Enter name for data set« den Namen, den R den eingelesenen Daten geben soll. Wählen Sie für mein Zahlenbeispiel den Namen Daten. Für R sind D und d unterschiedliche Buchstaben.
Mit »Variable names in file« sind die Namen der Spalten gemeint, in meiner Übungsdatei tdaten.csv Typ und Wert. Alle Spaltennamen sollten in der ersten Zeile stehen. Die Voreinstellung (Checkbox markiert) besagt, dass R die in der Datei vorhandenen Spaltennamen als Spaltennamen interpretieren soll (und nicht als Daten).
Die markierte Checkbox »Adjust for different column numbers« betrifft Dateien, deren Zeilen nicht alle die gleiche Anzahl von Einträgen aufweisen. Mit der Voreinstellung (Checkbox markiert) stellen Sie sicher, dass R alle Zeilen vollständig liest. R ermittelt die Spaltenzahl bei dieser Einstellung anhand der Zeile mit den meisten Einträgen.
Die standardmäßig markierte Option »Apply NA for blank cells in character variables« besagt, dass R eine leere Zelle als eine Zelle mit fehlendem Wert ansehen soll. R gibt einer solchen Zelle den Wert NA (not available). Sie sehen dies, wenn Sie Datensätze mit fehlenden Werten importieren und anschließend im Viewer betrachten (Menüleiste / Button Datenmatrix betrachten).
Das Feld «Characters indicating NA cells” betrifft Datensätze, in denen fehlende Werte durch eine bestimmte Zeichenkombination gekennzeichnet sind. Sie haben zum Beispiel einen Datensatz, in dem Zellen mit fehlenden Werten jeweils ein Fragezeichen enthalten. In diesem Fall schreiben Sie ein Fragezeichen in das Feld. R würde dann eine Zelle mit einem Fragezeichen als eine Zelle mit einem fehlenden Wert interpretieren.
Wählen Sie in der Rubrik »Location of Data File« die Option Internet URL, in der Rubrik »Field Separator« die Option Other (Specify: ;) und in der Rubrik »Decimal-Point Character« die Option Comma. Klicken Sie auf den OK-Button.
Schreiben Sie in das Feld »Internet URL« die folgende Adresse:
http://www.ghostwriting-service.de/misc/tdaten.csv
Klicken Sie wieder auf den OK-Button.
Prüfen Sie, ob der Import geklappt hat:
1. Betrachten Sie das untere Fenster (»Meldungen«): Sie sollten hier den Hinweis sehen, dass die Datenmatrix Daten 90 Zeilen und zwei Spalten hat. (Die CSV-Datei enthält 90 Beobachtungswerte für zwei Variablen.)
2. Betrachten Sie das Feld Datenmatrix in der Menüleiste: Hier muss Daten stehen, was bedeutet, dass sich alle weiteren Operationen auf den Datensatz Daten beziehen.
3. Betrachten Sie den Datensatz kurz im Viewer, Button Datenmatrix betrachten.
Sie können den geladenen Datensatz im RData-Format auf Ihrer Festplatte speichern. Hierzu wählen Sie im R Commander den Menüpunkt Aktive Datenmatrix / Save or export active data set / Save active data set.
Zum Öffnen einer solchen Datei haben Sie im R Commander den Menüpunkt Datei / Load data set. Falls die gesuchte RData-Datei im hierdurch geöffneten Dialog nicht angezeigt wird, wählen Sie im Dropdown-Menü rechts neben dem Feld für den Dateinamen die Option »Alle Dateien (*.*)«.
Das RData-Format ist ideal, wenn Sie Daten vorübergehend speichern wollen und hierfür ein Format wünschen, das R sehr leicht lesen kann.
Für die Archivierung von Daten wäre das RData-Format schlecht, weil R und das RData-Format weiterentwickelt werden. Sie müssen damit rechnen, dass zukünftige Versionen von R alte RData-Dateien nicht mehr lesen können.
Zur Archivierung eignet sich ein Textformat wie CSV.
Hierzu wählen Sie im R Commander den Menüpunkt Aktive Datenmatrix / Save or export active data set / Export active data set (Text). Sie speichern auf diese Weise (mit den Grundeinstellungen) eine Datei mit der Endung ».txt«. Diese Datei ist eine CSV-Datei mit durch Kommata getrennten Spalten und Punkten als Dezimaltrennzeichen.
Deskriptive Auswertung
Kennzahlen
Ausgangspunkt ist der importierte Datensatz Daten (im Feld Datenmatrix muss Daten stehen). Wählen Sie im R Commander den Menüpunkt Statistical analysis / Continuous variables / Numerical summaries:
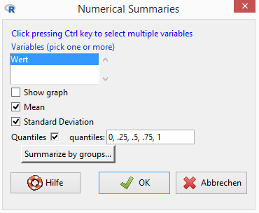
Der Datensatz Daten hat nur eine numerische Variable, die Variable Wert. R kann die angegebenen Kennzahlen (Mittelwert, Standardabweichung, fünf Quantile) daher nur für diese Variable berechnen.
Das 0-Quantil ist das Minimum, das 1-Quantil das Maximum, das 0,5-Quantil der Median (50 Prozent der Zahlen in der Spalte Wert sind höchstens so groß wie der Median).
Der Datensatz Daten hat die Spalte Typ mit den Kategorien A, B und C. Über den Button »Summarise by groups…« könnten Sie die Kennzahlen für jede dieser Kategorien separat ermitteln. Lassen Sie alle Einstellungen wie sie sind und klicken Sie auf den OK-Button.
Sie sehen im oberen Fenster (Registerkarte R Skript), wie der R Commander Ihre Eingaben im Dialogfeld »Numerical Summaries« in die Programmiersprache R übersetzt hat (ab der Zeile #####Numerical summaries#####). Diese Befehlsfolge finden Sie noch einmal im Fenster »Ausgabe«, gefolgt vom Ergebnis.
Das Menü Statistical analysis gehört zum Plugin EZR. Die Originalversion des R Commanders bietet weitere Möglichkeiten.
1. Gehen Sie zum Menüpunkt Original menu / Statistik / Deskriptive Statistik / Aktive Datenmatrix. Sie erhalten hierdurch eine Kurzbeschreibung aller im Datensatz enthaltenen Variablen. Für die Variable Typ ist für jede der drei Kategorien die Anzahl der Beobachtungswerte angegeben.
2. Gehen Sie zum Menüpunkt Original menu / Statistik / Deskriptive Statistik / Zusammenfassungen numerischer Variablen …:
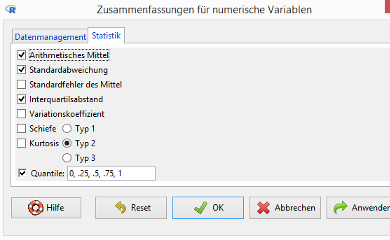
Sie können hier Kennzahlen wählen, die über das Angebot von EZR im Menü Statistical Analysis hinausgehen.
3. Gehen Sie zum Menüpunkt Original menu / Statistik / Deskriptive Statistik / Zähle die Anzahl der fehlenden Werte. Sie erhalten hierdurch für jede Spalte die Anzahl der NAs, das heißt die Anzahl der fehlenden Werte.
Grafiken
Wählen Sie im R Commander den Menüpunkt Original menu / Grafiken / Boxplot … Der hierdurch geöffnete Dialog Boxplot ist auf zwei Registerkarten aufgeteilt.
In der Registerkarte Datenmanagement legen Sie fest, für welche Variable Sie den Boxplot erstellen wollen. Bezogen auf den Datensatz Daten kann dies nur die Variable Wert sein, die daher bereits ausgewählt ist. Klicken Sie auf den Button »Grafik für die Gruppen…«. Als Gruppierungsvariable ist hier die Variable Typ ausgewählt. Bestätigen Sie dies, indem Sie im Fenster »Gruppen« auf den OK-Button klicken. In der Registerkarte Optionen können Sie alle Einstellungen lassen wie sie sind. Klicken Sie zum Abschluss auf den OK-Button des Dialogs »Boxplot«.
Der R Commander öffnet jetzt ein Grafikfenster:
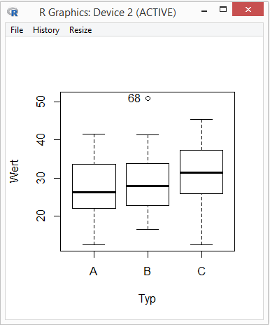
Zeile 68 im Datensatz ist laut Boxplot ein Ausreißer. Die drei Verteilungen (Typ A, B und C jeweils isoliert betrachtet) sind etwas schief.
Über das Menü File des Grafikfensters können Sie die Grafik speichern oder in die Zwischenablage kopieren. Hierbei ergeben sich die Breite und die Höhe der Grafik aus der Breite und der Höhe des Grafikfensters. Sie würden das Ergebnis daher beeinflussen, wenn Sie die Größe des Grafikfensters vor dem Speichern oder Kopieren durch Ziehen mit der Maus anpassen.
Erstellen Sie den Boxplot mit EZR. Wählen Sie hierzu den Menüpunkt Graphs and tables / Boxplot:
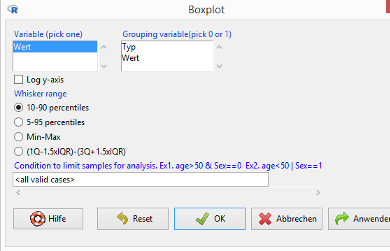
Wählen Sie als Gruppierungsvariable wieder Typ und als Whisker range »(1Q-1.5…)«. Klicken Sie auf den OK-Button.
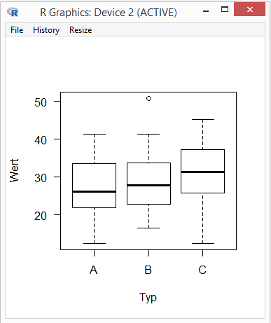
Es gibt in R zwei bedeutende Grafiksysteme: Base Graphics und Grid Graphics. Die Grundversion des R Commanders und EZR verwenden Base Graphics.
In Base Graphics sind zahlreiche Grafikmerkmale durch so genannte Grafikparameter festgelegt. R hat für alle Grafikparameter Grundeinstellungen, die nach dem Start automatisch gelten.
Zu den Grafikparametern gehört unter anderem die Ausrichtung der Zahlen oder Buchstaben an den Achsen. Die Grundeinstellung dieses Grafikparameters sehen Sie im ersten Boxplot: parallel zur jeweiligen Achse.
EZR hat diese Einstellung geändert: Im zweiten Boxplot sind die Zahlen an der Y-Achse horizontal ausgerichtet. Die neue Einstellung bleibt so lange wirksam, bis Sie sie zurücknehmen oder R beenden.
Wählen Sie noch einmal den Menüpunkt Original menu / Grafiken / Boxplot … und erstellen Sie den ersten Boxplot neu. Die Zahlen an der Y-Achse sind jetzt ebenfalls horizontal ausgerichtet.
EZR bietet den Menüpunkt Graphs and tables/ Graph settings, wo Sie vier Grafikparameter einstellen können: Line width (Breite der Linien), Axis label style (Ausrichtung der Zahlen oder Buchstaben an den Achsen), Font (Schriftfamilie), Font size (Schriftgröße). Wenn Sie auf den OK-Button klicken, sehen Sie eine Testgrafik mit den gewählten Einstellungen.
Das Plugin KMggplot2 verwendet ggplot2 und damit Grid Graphics. Grafikparameter aus Base Graphics sind daher für KMggplot2 bedeutungslos.
Gehen Sie zum Menüpunkt KMggplot2 / Box plot / Violin plot / Confidence interval…:
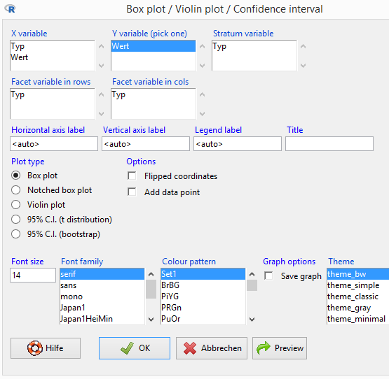
Markieren Sie in den Feldern X variable und Stratum variable jeweils den Eintrag Typ. Schreiben Sie in das Title-Feld »Boxplot mit Legende«. Klicken Sie dann auf den Preview-Button, um die gewählten Einstellungen zu testen.
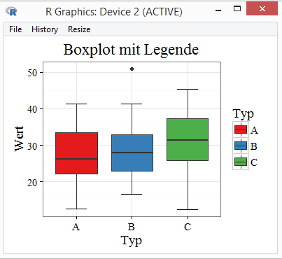
Sie merken durch Ausprobieren, dass die Felder Stratum variable und Colour pattern die Farbgestaltung steuern. Die Felder Facet variable in rows und Facet variable in cols wären für meinen Datensatz sinnvoll, wenn es neben Typ noch eine weitere Kategorie gäbe, zum Beispiel Region mit den Werten Bayern und Sachsen. Sie könnten dann einen Boxplot mit zwei Panels erstellen, im ersten Panel der Boxplot mit Typ A, B und C für Bayern und im zweiten Panel der gleiche Boxplot für Sachsen.
Über das Auswahlfeld Theme können Sie die Grafik in stilistischer Hinsicht anpassen, zum Beispiel an eine Grafik, die Sie mit EZR erstellt haben oder mit der Grundversion des R Commanders (theme_base oder ein ähnliches Theme).
Datensätze filtern
Gehen Sie im R Commander zum Menüpunkt Aktive Datenmatrix / Rows / Create subset data set:
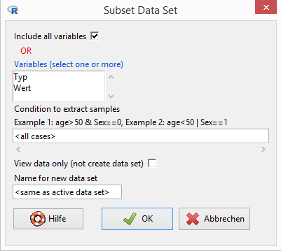
Schreiben Sie in das Feld Condition to extract samples:
Typ=="A"
Wählen Sie als Namen für den neuen Datensatz Daten_A und klicken Sie auf den OK-Button.
Im Feld Datenmatrix der Menüleiste steht jetzt Daten_A. Somit ist Daten_A der Datensatz, auf den sich alle weiteren Operationen beziehen. Durch einen Klick auf den Button »Datenmatrix betrachten« merken Sie, dass Daten_A nur noch Beobachtungswerte für Typ A enthält.
R deutet ein einfaches Gleichheitszeichen im Sinne einer Zuweisung. Mit X=3 geben Sie X den Wert 3. Im Beispiel soll R für jede Zeile des Datensatzes Daten prüfen, ob in der Spalte Typ ein A steht. Eine solche Prüfung veranlassen Sie mit einem doppelten Gleichheitszeichen.
Die Anweisung Typ==A hätte eine Fehlermeldung verursacht, weil mit A auch eine Variable gemeint sein könnte. Durch die Anführungszeichen ("A") stellen Sie klar, dass der Buchstabe A im Sinne eines Symbols zu verstehen ist. Möglich sind auch einfache Anführungszeichen ('A').
Interessant ist die im Dialog »Subset Data Set« in Example 1 enthaltene Anweisung Sex==0. R deutet die Null hier als Zahl (Namen von Variablen dürfen in R nicht mit Ziffern beginnen). Mit "0" wäre nicht die Zahl Null gemeint, sondern das entsprechende Symbol. Die Anweisung Sex=="0" wäre für eine Spalte mit Zahlen unsinnig.
Wählen Sie den Menüpunkt Aktive Datenmatrix / Variables / Show variables in active data set. Sie sehen jetzt im Fenster Ausgabe, dass es die Typen "A", "B" und "C" gibt und die Spalte Wert Zahlen enthält. Sie finden hier die korrekte Schreibweise: Werte aus der Typ-Spalte mit Anführungszeichen und Werte aus der Wert-Spalte ohne.
Es kann beim Import von Daten vorkommen, dass R die in einer Spalte enthaltenen Zahlen als Symbole auffasst. Für solche Fälle haben Sie den Menüpunkt Aktive Datenmatrix / Variables / Convert factors or character variables of numeric data to numeric variables.
Neben == kennt R auch != (ungleich), < (kleiner als), <= (kleiner oder gleich), > (größer als) und >= (größer oder gleich).
Klicken Sie in der Menüleiste auf das Feld Datenmatrix und wählen Sie den Datensatz Daten.
Gehen Sie anschließend wieder zum Menüpunkt Aktive Datenmatrix / Rows / Create subset data set. Schreiben Sie in das Feld Condition to extract samples:
Typ=="B" & Wert>=35.03
Markieren Sie die Checkbox View data only (not create data set) und klicken Sie auf den OK-Button.
Sie sehen das Ergebnis jetzt im Fenster Ausgabe des R Commanders:
| Typ | Wert | |
|---|---|---|
| 25 | B | 35.10608 |
| 67 | B | 41.29943 |
| 68 | B | 50.91078 |
| 72 | B | 36.24275 |
| 81 | B | 35.84405 |
| 86 | B | 40.06877 |
In Typ=="B" & Wert>=35.03 bedeutet das &-Symbol, dass beide Bedingungen erfüllt sein müssen. Gesucht sind alle Zeilen mit einem B in der Spalte Typ und einer Zahl größer oder gleich 35,03 in der Spalte Wert. R akzeptiert für Zahlen nur die englische Schreibweise (35.03 statt 35,03).
Ein senkrechter Strich ist im Sinne von und/oder gemeint:
Typ=="B" | Wert>=35.03 liefert alle Zeilen, die eine oder beide Bedingungen erfüllen.
Neue Variablen bilden
Stellen Sie sicher, dass im Feld Datenmatrix der Menüleiste der Name Daten steht. Wählen Sie den Menüpunkt Aktive Datenmatrix / Variables / Create new variable:
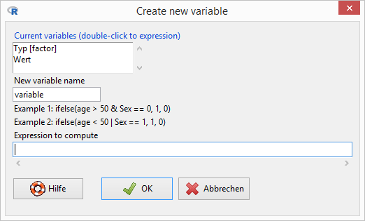
Schreiben Sie in das Feld »New variable name« Wert_cm und in das Feld Expression to compute
Wert*10
Klicken Sie auf den OK-Button.
Der Datensatz Daten enthält jetzt neben den ursprünglichen Variablen (Typ, Wert) die Variable Wert_cm (Menüleiste, Button Datenmatrix betrachten).
Die Operatoren +, -, * und / stehen für die vier Grundrechenarten. 2^3 ist 2 hoch drei und 2^(1/3) ist dritte Wurzel aus 2. Mit abs(-2) berechnen Sie den Betrag von -2 und mit log(3) den natürlichen Logarithmus von 3. Runde Klammern dienen dazu, Anweisungen zu gruppieren, zum Beispiel
(3+4)/12
In Excel gibt es die Funktion WENN(), die drei Argumente hat:
- Eine Bedingung, die geprüft werden soll
- Das Ergebnis, falls die Bedingung erfüllt ist
- Das Ergebnis, falls die Bedingung nicht erfüllt ist
In R heißt die entsprechende Funktion ifelse().
Wählen Sie wieder den Menüpunkt Aktive Datenmatrix / Variables / Create new variable. Schreiben Sie in das Feld »New variable name« Typ2 und in das Feld Expression to compute
ifelse(Typ=="A"|Typ=="B","AB","C")
Klicken Sie auf den OK-Button.
Der Datensatz hat jetzt Typ2 als zusätzliche Spalte.
Die mit der Funktion ifelse() gegebene Anweisung ist zeilenweise gemeint. Beginnen Sie mit der ersten Zeile. Sie prüfen, ob hier in der Typ-Spalte ein A oder ein B steht. Ist diese Bedingung erfüllt, so schreiben Sie in die Typ2-Spalte die Zeichenfolge AB. Andernfalls schreiben Sie in die Typ2-Spalte ein C.
Sie wiederholen das Verfahren für die zweite Zeile, dann für die dritte Zeile und so weiter. Aus drei Kategorien (A, B, C) haben Sie dann zwei gemacht (AB, C).
Wählen Sie noch einmal den Menüpunkt Aktive Datenmatrix / Variables / Create new variable. Schreiben Sie in das Feld »New variable name« Wert_K und in das Feld Expression to compute
ifelse(Wert<22.97, "Niedrig",ifelse(Wert>35.03, "Hoch","Mittel"))
Klicken Sie auf den OK-Button.
Dies ist ein Beispiel für eine verschachtelte Anweisung, die auch in Excel möglich wäre. Für die erste Zeile würde die Anweisung bedeuten:
- Zahl in der Wert-Spalte kleiner als 22,97 - Niedrig in die Wert_K-Spalte eintragen
- Zahl in der Wert-Spalte größer als 35,03 - Hoch in die Wert_K-Spalte eintragen
- Keine der beiden Bedingungen erfüllt - Mittel in die Wert_K-Spalte eintragen
Gehen Sie zum Menüpunkt Aktive Datenmatrix / Variables / Bin numeric variables:
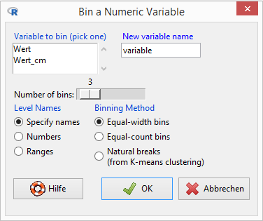
Markieren Sie im Feld Variable to bin die Variable Wert, schreiben Sie in das Feld New variable name Wertklassen, schieben Sie den Regler Number of bins auf 6, wählen Sie bei den Level Names die Option Numbers und klicken Sie auf den OK-Button.
Der Datensatz hat jetzt die zusätzliche Variable Wertklassen, die den Zahlen in der Spalte Wert Ratings zuordnet; 1 für die niedrigsten Zahlen in der Spalte Wert, 6 für die höchsten.
Gehen Sie zum Menüpunkt Aktive Datenmatrix / Variables / Show variables in active data set. Sie sehen jetzt, dass die Ziffern 1 bis 6 der Variablen Wertklassen keine Zahlen sind. Die Ziffern 1 bis 6 dienen hier als Symbole.
Gehen Sie zum Menüpunkt Aktive Datenmatrix / Variables / Convert factors or character variables of numeric data to numeric variables. Markieren Sie die Variable Wertklassen und klicken Sie auf den OK-Button. Stimmen Sie der Frage zu, ob die Variable überschrieben werden soll.
Gehen Sie jetzt noch einmal zum Menüpunkt Aktive Datenmatrix / Variables / Show variables in active data set. Die Ziffern 1 bis 6 sind jetzt Zahlen. Testen Sie das. Gehen Sie zum Menüpunkt Original menu / Statistik / Deskriptive Statistik / Aktive Datenmatrix.
Mittelwertvergleiche
Ich möchte für den Datensatz Daten die Mittelwerte der drei Typen vergleichen. Hierzu will ich zunächst testen, ob meine Daten mit zwei Modellannahmen im Einklang stehen.
Die erste Modellannahme besagt, dass die Beobachtungswerte für die drei Typen jeweils einer normalverteilten Grundgesamtheit entstammen.
Für die drei Tests auf Normalverteilung könnte ich auf Basis meines Datensatzes drei neue Datensätze erstellen, einen Datensatz mit den Werten für Typ A, einen Datensatz mit den Werten für Typ B und einen Datensatz mit den Werten für Typ C. Ich hätte den Datensatz Daten (90 Beobachtungswerte) dann in drei Datensätze mit jeweils 30 Beobachtungswerten aufgespalten.
Diese Vorgehensweise wäre umständlich. Beim Test auf Normalverteilung (und vielen anderen Verfahren) besteht daher die Möglichkeit, die Untersuchung auf Teile des verwendeten Datensatzes zu beschränken.
Wählen Sie im R Commander den Menüpunkt Statistical analysis / Continuous variables / Kolmogorov-Smirnov test for normal distribution:
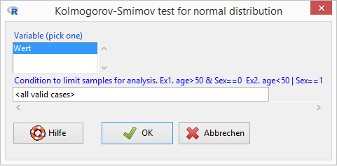
Schreiben Sie in das Feld Condition to limit samples for analysis
Typ=="A"
Klicken Sie auf den OK-Button.
Sie sehen jetzt im Feld Ausgabe des R Commanders, dass R zwei Tests durchgeführt hat, einen Kolmogorov-Smirnov-Test und einen Shapiro-Wilk-Test. Die Nullhypothese HO »Die Daten stammen aus einer normalverteilten Grundgesamtheit« wird bei beiden Tests angenommen (p-Werte größer als 0,05). Die zugehörige Grafik zeigt, dass diese Einschätzung plausibel ist.
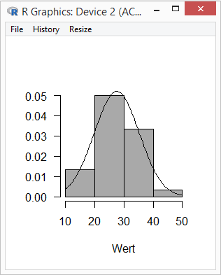
Für Typ B und C erhalte ich ähnliche Ergebnisse.
Die zweite Modellannahme besagt, dass die drei Grundgesamtheiten, bezogen auf die Variable Wert, die gleiche Varianz haben.
Wählen Sie den Menüpunkt Original menu / Statistik / Varianzen / Levene-Test …:
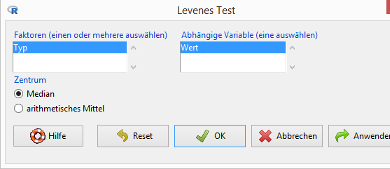
Im Feld Faktoren bedeutet der markierte Eintrag Typ, dass die Werte der Variablen Typ die Gruppeneinteilung definieren. Wählen Sie als Zentrum die Option arithmetisches Mittel und klicken Sie auf den OK-Button.
Das Signifikanzniveau p= 0.3242 spricht für die Annahme der Varianzgleichheit zwischen den drei Gruppen.
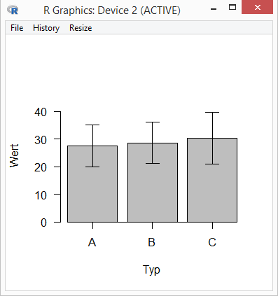
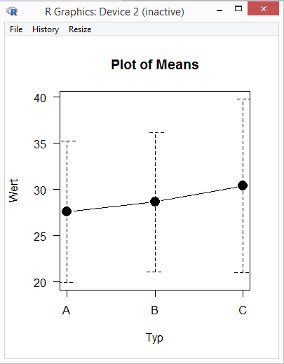
Gehen Sie zum Menüpunkt Graphs and tables / Bar graph (Means). Mit Typ als Grouping variable erhalten Sie eine Grafik mit den Mittelwerten und Standardabweichungen der drei Gruppen.
Gehen Sie zum Menüpunkt Statistical analysis / Continuous variables / One-way ANOVA:
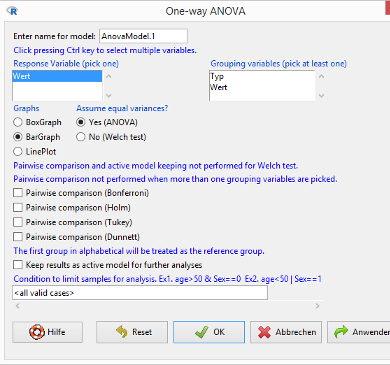
Wählen Sie als Grouping variable Typ, in der Rubrik Graphs LinePlot und als Methode Pairwise comparison (Tukey). Klicken Sie dann auf den OK-Button.
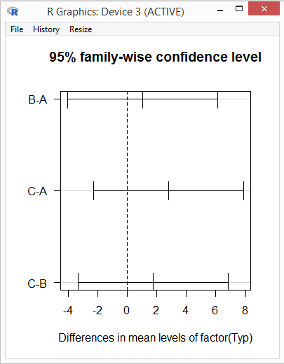
Ergebnis (Fenster Ausgabe des R Commander):
| diff | lwr | upr | p adj | |
|---|---|---|---|---|
| B-A | 1.041836 | -4.038178 | 6.121849 | 0.8767100 |
| C-A | 2.806970 | -2.273043 | 7.886984 | 0.3893288 |
| C-B | 1.765135 | -3.314879 | 6.845148 | 0.6864105 |
Die linke Spalte zeigt, für welche Gruppen die Mittelwerte jeweils verglichen worden sind. Keiner dieser drei Vergleiche hat ein signifikantes Ergebnis geliefert (alle drei p-Werte sind größer als 0,05).
R hat für diesen Test zwei Plots erstellt (in zwei übereinander liegenden Fenstern):
Regression (Ordinary Least Squares)
Gehen Sie im R Commander zum Menüpunkt Datei / Read data set from attached package:
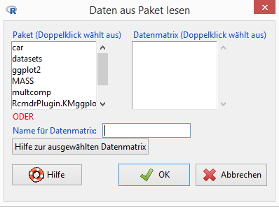
Markieren Sie im Feld Paket per Doppelklick den Eintrag car (Companion to Applied Regression). Wählen Sie jetzt im Feld Datenmatrix per Doppelklick den Eintrag Prestige. Klicken Sie anschließend auf den OK-Button.
Im Feld Datenmatrix der Menüleiste steht jetzt Prestige.
Die erste Regressionsgleichung:
(1) income = α + β1prestige + β2women + ε
Gehen Sie zum Menüpunkt Statistical analysis / Continuous variables / Linear regression:
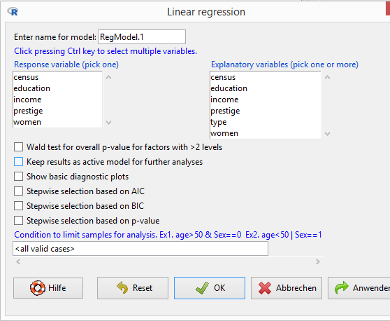
Schreiben Sie in das Feld »Enter name for model« reg_1. Wählen Sie als Response variable income und als Explanatory variables prestige und women. Markieren Sie die Optionen «Keep results as active model for further analyses” und «Show basic diagnostic plots”. Klicken Sie zum Abschluss auf den OK-Button.
Sie sehen jetzt in der Menüleiste, dass reg_1 im Feld Modell steht. Sie haben hierdurch die Möglichkeit, das Modell reg_1 weiter zu untersuchen.
Die Schätzergebnisse finden Sie im Fenster Ausgabe. Danach sind die Einflüsse der Variablen prestige und women signifikant. Multikollinearität kann verneint werden, beide VIF-Werte liegen bei eins.
Der erste Diagnoseplot (Residuals vs Fitted) deutet darauf hin, dass die Modellannahme der Homoskedastizität verletzt ist. Ich prüfe dies mit einem Breusch-Pagan-Test.
Wählen Sie den Menüpunkt Original menu / Modelle / Numerische Diagnose / Breusch-Pagan Test auf Heteroskedastizität …:
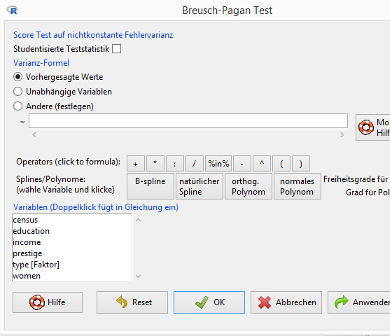
Lassen Sie die Einstellungen wie vorgeschlagen (Varianz-Formel = Vorhergesagte Werte) und klicken Sie auf den OK-Button.
Die Nullhypothese H0 des Breusch-Pagan-Tests lautet: Die Bedingung der Homoskedastizität ist für das untersuchte Modell erfüllt. Für ein brauchbares Regressionsmodell sollte der p-Wert daher größer als 0,05 sein. Das ist hier jedoch nicht der Fall (p-value < 2.2e-16).
Das Regressionsmodell reg_1 ist daher inakzeptabel.
Möglicherweise finden Sie im Fenster Meldungen des R Commanders eine rot hervorgehobene Meldung von der Art »FEHLER: there is no package called 'aod'«. Damit ist gemeint, dass R auf Funktionen aus dem Package aod zurückgreifen wollte und dies nicht konnte.
Wenn eine solche Meldung angezeigt wird, dann sollten Sie das Problem beseitigen. Gehen Sie in einem solchen Fall in der R-Konsole zum Menüpunkt Packages / Load package … Prüfen Sie dort, ob das Package im Auswahlfeld enthalten ist. Wenn ja: Markieren Sie das Package und klicken Sie auf den OK-Button.
Andernfalls müssten Sie das Package zunächst installieren (R-Konsole, Menüpunkt Packages / Install package(s) …) und dann laden (R-Konsole, Menüpunkt Packages / Load package …).
Wählen Sie im R Commander den Menüpunkt Original menu / Statistik / Modelle anpassen / Lineares Regressionsmodell …:
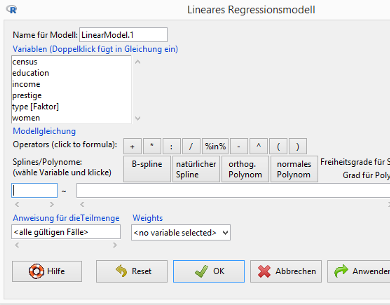
Sie haben hier die Möglichkeit, die Regressionsgleichung zu spezifizieren. Die unbrauchbare Regressionsgleichung
(1) income = α + β1prestige + β2women + ε
lautet in der R-Syntax für Regressionsgleichungen
income ~ prestige + women
Sie müssten in das Feld vor der Tilde income eintragen und in das Feld nach der Tilde prestige+women (income ~ prestige + women).
Schätzen Sie folgende Regressionsgleichung:
(2) log(income) = α + β1prestige-1 + β2women2 + ε
Sie könnten hierzu den Datensatz Prestige um drei neue Variablen erweitern:
- income_log = log(income)
- prestige_rez = 1/prestige
- women_quad = women^2
Sie hätten dann als zu schätzende Regressionsgleichung:
(2a) income_log = α + β1prestige_rez + β2women_quad + ε
Eine einfachere Lösung besteht darin, die drei Transformationen in der Regressionsformel zu spezifizieren. Hierbei ist wichtig, dass alle Angaben eindeutig sind. Ein Symbol wie + kann im Sinne einer Berechnung gemeint sein oder im Sinne der Formelsyntax. Statt 1+1 gibt es daher auch den Ausdruck I(1+1), durch den klargestellt wird, dass 1+1 im Sinne einer Berechnung gemeint ist; I wie der erste Buchstabe des Worts Italien.
Gleichung (2) lautet in der R-Formelsyntax:
log(income) ~ I(1/prestige)+I(women^2)
Durch l(1/prestige) wird unmissverständlich klargestellt, dass die erste erklärende Variable aus der Variablen prestige abgeleitet ist, für jede Zahl x in der prestige-Spalte durch die Formel 1/x. Entsprechendes gilt für I(women^2).
Schätzen Sie die Regressionsgleichung, indem Sie log(income) in das Feld vor der Tilde und I(1/prestige)+I(women^2) in das Feld nach der Tilde eingeben. Geben Sie dem Modell den Namen reg_2 und klicken Sie auf den OK-Button.
Alle drei Koeffizienten sind signifikant (p-Wert jeweils < 2e-16).
Im Feld Modell der Menüleiste steht reg_2. Gehen Sie zum Menüpunkt Original menu / Modelle / Grafiken / Grundlegende diagnostische Grafiken. Sie sehen jetzt die vier Diagnoseplots. Der erste Plot (Residuals vs Fitted) macht einen besseren Eindruck als beim Modell reg_1.
Wählen Sie wieder den Menüpunkt Original menu / Modelle / Numerische Diagnose / Breusch-Pagan Test auf Heteroskedastizität …, lassen Sie die Einstellungen unverändert und klicken Sie auf den OK-Button. Der p-Wert beträgt jetzt 0.1527 (Modellannahme der Homoskedastizität laut Breusch-Pagan-Test erfüllt).
Der zweite Diagnoseplot (Normal Q-Q) deutet darauf hin, dass die Residuen nicht normalverteilt sind. Gehen Sie daher zum Menüpunkt Original menu / Modelle / Füge Regressionsstatistiken zu den Daten hinzu …:
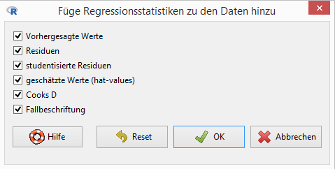
Markieren Sie hier nur die Checkbox Residuen. Klicken Sie dann auf den OK-Button.
Der Datensatz Prestige hat jetzt residuals.reg_2 als neue Spalte.
Das lineare Regressionsmodell (Ordinary Least Squares) setzt für die Residuen drei Modellannahmen voraus:
- Die Residuen sind jeweils normalverteilt mit Erwartungswert 0
- Die Residuen haben alle die gleiche Varianz (Homoskedastizität)
- Die Residuen sind voneinander stochastisch unabhängig
Wenn ich von diesen Modellannahmen ausgehe, dann macht es Sinn, den Vektor der Residuen so zu behandeln, als stamme er von einer einzigen normalverteilten Zufallsvariablen. Unter den Modellannahmen wäre zu erwarten, dass der Residuenvektor einen Kolmogorov-Smirnov- oder Shapiro-Wilk-Test besteht.
Gehen Sie zum Menüpunkt Statistical analysis / Continuous variables / Kolmogorov-Smirnov test for normal distribution. Machen Sie den Test für residuals.reg_2.
Gemäß Kolmogorov-Smirnov-Test sind die Residuen normalverteilt (p-Wert 0,3231), gemäß Shapiro-Wilk-Test nicht (p-Wert 0,0005769). Dies spricht gegen das Modell reg_2.
Grafiken speichern und anpassen
Eine Grafik in der gewünschten Größe speichern
Wählen Sie im R Commander den Menüpunkt Graphs and tables / Graph settings und dort Graph size = Medium und Graph shape = Square. Klicken Sie auf den OK-Button.
Sie sehen jetzt eine Grafik, die diese Einstellungen veranschaulicht. Außerdem finden Sie im oberen Fenster des R Commenders (Registerkarte R Skript) die Zeile
windows(width=7, height=7)
Lassen Sie die Größe des Grafikfensters unverändert. Wählen Sie im Grafikfenster den Menüpunkt File / Save as / Png … und speichern Sie die Grafik. Die Grafik ist 672 Pixel breit und 672 Pixel hoch (die Grafikdatei mit der rechten Maustaste anklicken, Kontextmenü Eigenschaften).
Angenommen, Ihre Grafik soll 390 Pixel breit und 310 Pixel hoch sein. Sie rechnen hierzu zwei Dreisätze, die ich in abgekürzter Form wiedergebe:
width=7 ergibt 672 Pixel Breite
width=7*390/672 ergibt 390 Pixel Breite
height=7 ergibt 672 Pixel Höhe
height=7*310/672 ergibt 310 Pixel Höhe
Schreiben Sie in das obere Fenster des R Commanders (Registerkarte R Skript):
windows(width=7*390/672, height=7*310/672)
Markieren Sie diese Anweisung mit der Maus und klicken Sie auf den Button »Befehl ausführen« unterhalb des Fensters. Sie sehen jetzt ein leeres Grafikfenster.
Gehen Sie mit dem Cursor in folgende Zeile:
plot(sin, xlim=c(0,10), main="Sample")
Klicken Sie wieder auf den Button Befehl ausführen. Lassen Sie die Größe des Grafikfensters unverändert, wählen Sie im Grafikfenster den Menüpunkt File / Save as / Png … und speichern Sie die Grafik. Die neue Grafik ist 390 Pixel breit und 310 Pixel hoch.
Grafikparameter anpassen (Base Graphics)
Ich verwende wieder den Datensatz Prestige (Menüpunkt Datei / Read data set from attached package, dort car im Paket-Feld auswählen und dann als Datenmatrix Prestige).
Gehen Sie im R Commander zum Menüpunkt KMggplot2 / Scatter plot… und erstellen Sie eine Grafik mit folgenden Einstellungen:
- X variable: income
- Y variable: prestige
- Title: Scatterplot
- Smoothing with C.I. (linear regression)
- Font size: 12
- Font family: sans
- Theme: theme_few
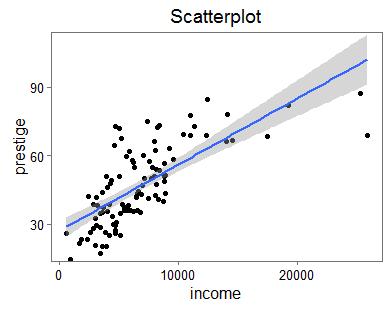
Angenommen, Sie wollen alle Grafiken so gestalten, dass Sie zu diesem Scatterplot passen. Solange Sie mit KMggplot2 arbeiten ist klar, wie Sie dies erreichen (theme_few wählen).
Gehen Sie zum Menüpunkt Graphs and tables / Bargraph (Means) und erstellen Sie den folgenden Plot (Response: income, Grouping variable: type):
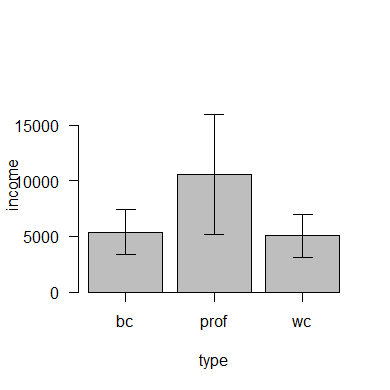
Die von EZR bewirkte horizontale Ausrichtung der Y-Achsenbeschriftung hat hier zur Folge, dass für den Achsentitel income zu wenig Platz ist.
Wie können Sie diese Grafik an den Scatterplot anpassen?
Den Ausgangspunkt bilden die im oberen Fenster des R Commanders (Registerkarte R Skript) enthaltenen Anweisungen, die Sie über den Menüpunkt Graphs and tables / Bargraph (Means) an R gesendet haben.
Von Interesse sind die folgenden Zeilen:
barx <- barplot(bar.means, ylim=c(ifelse(min(bar.means, na.rm=TRUE)>0, 0, min(bar.means-bar.sds, na.rm=TRUE)*1.2), max(bar.means+bar.sds, na.rm=TRUE)*1.2), xlab="type", ylab="income", axis.lty=1)
error.bar(barx, bar.means, bar.sds)
Kopieren Sie diese Anweisungen und fügen Sie diese Anweisungen noch einmal am Ende des Fensters ein. Der neue Plot soll 390 Pixel breit und 390 Pixel hoch werden. Die hierfür erforderliche Anweisung lautet windows(width=7*390/672, height=7*390/672). Ich hatte dies im letzten Abschnitt erläutert.
Der neue R-Code sieht dann folgendermaßen aus:
windows(width=7*390/672, height=7*390/672)
barx <- barplot(bar.means, ylim=c(ifelse(min(bar.means, na.rm=TRUE)>0, 0, min(bar.means-bar.sds, na.rm=TRUE)*1.2), max(bar.means+bar.sds, na.rm=TRUE)*1.2), xlab="type", ylab="income", axis.lty=1)
error.bar(barx, bar.means, bar.sds)
Markieren Sie hiervon mit der Maus alles außer der letzten Zeile und klicken Sie auf den Button Befehl ausführen. Sie sehen jetzt einen Barplot, den die Funktion barplot() erstellt hat. Der Barplot ist eine eigenständige Grafik, die Sie als Grafikdatei speichern könnten.
Markieren Sie die letzte Zeile und klicken Sie wieder auf den Button Befehl ausführen. Sie sehen jetzt, dass der Barplot drei Error Bars erhalten hat. Die Funktion error.bar(), die diese Error Bars gezeichnet hat, wird als Low-level-Funktion bezeichnet. Eine Low-level-Funktion ergänzt eine bereits vorhandene Grafik.
Der gerade getestete R-Code funktioniert, weil Sie die Grafik zuvor über den Menüpunkt Graphs and tables / Bargraph (Means) erstellt hatten. R braucht für die Grafik die Werte der Variablen bar.means (Höhe der Balken) und bar.sds (Breite der Error Bars). Sie sehen in der Registerkarte R Skript unterhalb der Zeile #####Bar graph(Means)#####, dass R diese Werte berechnet hat.
Es gibt in Base Graphics sehr viele Low-level-Funktionen. Hierzu gehören:
- axis() - zeichnet und beschriftet eine Achse
- box() - zeichnet einen Rahmen um das Panel
- title() - erstellt einen Grafiktitel
- mtext() - schreibt Text in den Randbereich
High-level-Funktionen erstellen komplette Grafiken. Solche Funktionen haben den Nachteil, dass Sie die hiermit erzielten Ergebnisse nur begrenzt steuern können. Eine mögliche Lösung besteht darin, der betreffenden High-level-Funktion, hier barplot(), Aufgaben zu entziehen und diese Aufgaben mit Low-level-Funktionen zu erledigen.
Wichtig sind die folgenden Anweisungen:
- xlab="" - löscht den Titel der X-Achse
- ylab="" - löscht den Titel der Y-Achse
- main="" - löscht den Titel
- sub="" - löscht den Untertitel
- axes=FALSE - löscht die Achsen
Sie finden im R-Code für den Barplot die Anweisungen xlab="type"und ylab="income". Machen Sie daraus xlab="" und ylab="". Es kommt häufig vor, dass die Argumente xlab und ylab nicht spezifiziert sind und die High-level-Funktion trotzdem Achsentitel erstellt. In einem solchen Fall müssten Sie die Anweisungen xlab="" und ylab="" zu den Argumenten der High-level-Funktion hinzufügen.
Die Argumente main und sub sind für mein Beispiel bedeutungslos. Der Barplot hat weder einen Titel noch einen Untertitel, der sich am unteren Rand der Grafik befinden würde. Sie können daher auf die Angaben main="" und sub="" verzichten.
Das axes-Argument ist für den Barplot nicht spezifiziert. Sie müssen die Anweisung axes=FALSE daher bei den Argumenten von barplot() ergänzen. Alle Argumente müssen durch Kommata getrennt sein. Außerdem dürfen Sie ein Argument nur einmal spezifizieren; barplot(…, axes=TRUE, …, axes=FALSE) wäre unzulässig. Alles muss eindeutig sein.
Sie haben jetzt folgendes Zwischenergebnis:
windows(width=7*390/672, height=7*390/672)
barx <- barplot(bar.means, ylim=c(ifelse(min(bar.means, na.rm=TRUE)>0, 0, min(bar.means-bar.sds, na.rm=TRUE)*1.2), max(bar.means+bar.sds, na.rm=TRUE)*1.2), xlab="", ylab="", axis.lty=1, axes=FALSE)
error.bar(barx, bar.means, bar.sds)
Markieren Sie diese Anweisungen mit der Maus und klicken Sie auf den Button Befehl ausführen. Sie erhalten dann folgende Grafik:
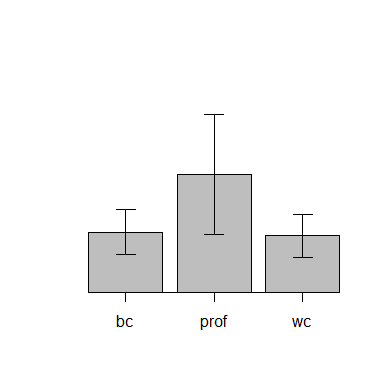
Der Barplot hat immer noch eine X-Achse, trotz der Anweisung axes=FALSE. Das - ungewöhnliche - Zwischenergebnis ist für meine Zwecke aber ausreichend, weil die Y-Achse gelöscht ist und ich daher in der Lage bin, beide Achsen unabhängig voneinander zu gestalten.
Ich beginne mit der X-Achse. Im Vergleich zum theme_few sind die Ticks zu lang, die Achsenbeschriftungen zu weit unten und die Buchstaben zu groß. Außerdem brauche ich graue Ticks.
Die Länge der Ticks wird durch den Parameter tcl gesteuert, der in der Grundeinstellung auf den Wert -0.5 gesetzt ist. Das Vorzeichen gibt hierbei an, auf welcher Seite der Achse die Ticks gezeichnet werden sollen (außerhalb oder innerhalb des Panels). Ich kürze die Ticks und setze daher par(tcl=-0.18).
Die Funktion par() kann alle von Base Graphics verwendeten Grafikparameter vorübergehend ändern (R startet immer mit den Grundeinstellungen). Eine mit par() festgelegte Abweichung von den Grundeinstellungen gilt während der laufenden Session für alle mit Base Graphics erstellten Grafiken. Die Einstellung par(tcl=-0.18) gilt für sämtliche Achsen, nicht nur für die X-Achse.
Ich halte es für sinnvoll, die für eine bestimmte Grafik mit par() geänderten Grafikparameter schnellstmöglich wieder auf ihre Grundeinstellungen zurückzusetzen. Für die Länge der Ticks wäre dies die Anweisung par(tcl=-0.5).
Die Größe der Achsenbeschriftungen wird durch den Parameter cex.axis gesteuert, mit 1 als Grundeinstellung. Der Wert dieses Parameters ist als Verhältnis zur Standardschriftgröße zu verstehen. Ich will hier die Achsenbeschriftung verkleinern und setze par(cex.axis=0.8).
Der Abstand der Achsenbeschriftungen von den Achsen wird durch den Parameter mpg gesteuert. Die Grundeinstellung lautet mgp= c(3,1,0). Die Schreibweise c(…) bedeutet, dass es sich um einen Vektor handelt. Alle Komponenten eines Vektors sind in R durch Kommata getrennt. Maßgeblich für den Abstand der Achsenbeschriftung von den Achsen ist der zweite Wert, den ich von 1 auf 0.2 reduziere.
Die Anweisung lautet entsprechend par(mgp=c(3,0.2,0)).
Base Graphics hat für die Farbe der Ticks keinen speziellen Parameter. Ich ändere daher die Farbe, in der R die Achsen, bestimmte Arten von Linien und Umrandungen zeichnet. Hierfür gibt es den Parameter fg, der in der Grundeinstellung gleich "black" ist. Ich setze par(fg="#666666") und nach dem Zeichnen des Barplots sofort wieder par(fg="black"), damit die Error Bars schwarz bleiben.
Ich komme jetzt zur Y-Achse, die ich mit der Anweisung axis(2) erstelle. Zwei steht hier für die Seite: 1 = unten, 2 = links, 3 = oben, 4 = rechts. Die Funktion axis() erstellt die Ticks und die Beschriftungen automatisch. Sie haben aber die Möglichkeit, dies manuell festzulegen, zum Beispiel axis(2, at=c(5000,10000,15000), labels=c("5","10","15")).
Außerdem können Sie axis() für eine Seite mehrfach nutzen, insbesondere um Achsen mit unterschiedlich langen Ticks zu erzeugen (zum Beispiel lange Ticks für runde Zahlen, kurze Ticks für Zwischenwerte).
In Base Graphics kann man Grafikparameter, für die dies sinnvoll erscheint in der Regel auch in Grafikfunktionen als Argumente spezifizieren.
Ich nutze diese Möglichkeit für drei Grafikparameter:
axis(2,tcl=-0.2,las=1,mgp=c(3,0.4,0))
Ich hatte gesetzt par(tcl=-0.18, mgp=c(3,0.2,0)). Dies ist jetzt für die Y-Achse unerheblich, weil die Argumente einer Grafikfunktion für diese Funktion eine höhere Priorität haben als mit par() spezifizierte Grafikparameter. Außerdem habe ich den Parameter las auf 1 gesetzt, was bedeutet, dass die Achsenbeschriftungen horizontal verlaufen sollen. Die Einstellung par(cex.axis=0.8) gilt auch für die Y-Achse, weil ich in axis() nichts anderes festgelegt habe.
Für die Farbe der Ticks hat axis() das Argument col.ticks, für das ich den Grauton #666666 angebe. Ich erhalte somit:
axis(2, tcl=-0.2, las=1, mgp=c(3,0.4,0), col.ticks="#666666")
Zum Abschluss der Achsengestaltung brauche ich einen Rahmen um das Panel, den ich mit box() erzeuge. Diese Funktion zeichnet in der Grundeinstellung schwarze durchgezogene Linien. Für einen grauen Rahmen schreibe ich box(col="#666666").
Mein Zwischenergebnis:
windows(width=7*390/672, height=7*390/672)
par(tcl=-0.18, cex.axis=0.8, mgp=c(3,0.2,0), fg="#666666")
barx <- barplot(bar.means, ylim=c(ifelse(min(bar.means, na.rm=TRUE)>0, 0, min(bar.means-bar.sds, na.rm=TRUE)*1.2), max(bar.means+bar.sds, na.rm=TRUE)*1.2), xlab="", ylab="", axis.lty=1, axes=FALSE)
par(fg="black")
error.bar(barx, bar.means, bar.sds)
axis(2, tcl=-0.2, las=1, mgp=c(3,0.4,0), col.ticks="#666666")
box(col="#666666")
par(tcl=-0.5, mgp=c(3,1,0), cex.axis=1)
Wichtig ist hier die Reihenfolge der Anweisungen:
- Erst öffne ich mit windows() ein Grafikfenster
- Dann setze ich Grafikparameter mit par()
- Dann verwende ich die High-level-Funktion barplot()
- Dann ändere ich die Linienfarbe, par(fg="black")
- Dann nutze ich Low-level-Funktionen
Weil ich box() nach barplot() und axis() ausführe, liegt der von box() gezeichnete Rahmen über den beiden Achsen. Zum Schluss habe ich die übrigen der mit par() gesetzten Grafikparameter auf ihre Grundeinstellungen zurückgesetzt.
An R übermittelte Anweisungen funktionieren nur, wenn sie fehlerfrei sind. Sollten Probleme auftreten, so können Sie den Code schrittweise durchgehen: Die erste Anweisung mit der Maus markieren und ausführen, wenn dies klappt die zweite Anweisung mit der Maus markieren und ausführen und so weiter.
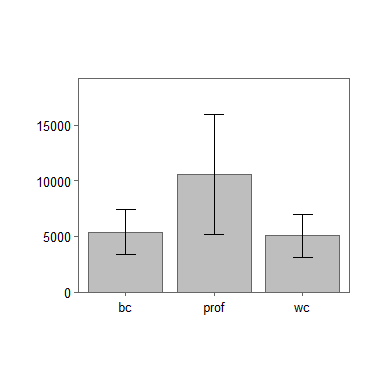
Im obigen Diagramm sind wegen par(fg="#666666") die Umrandungen der Balken grau. Ich kann die Balken trotzdem farblich gestalten.
Schreiben Sie im oberen Fenster des R Commanders (Registerkarte R Skript) in eine neue Zeile ?barplot, lassen Sie den Cursor in dieser Zeile und klicken Sie auf den Button Befehl ausführen. Ihr Standardbrowser öffnet jetzt die Hilfe zur Funktion barplot().
In der Rubrik Arguments finde ich die Argumente col für die Balkenfarbe und border für die Umrandungen der Balken. Ich setze col="#ffe5cc" und border="black". Es wäre auch möglich, für jeden der drei Balken separate Angaben zu machen, zum Beispiel col=c("#ffe5cc", "red", "blue").
Für den Titel verwende ich die Funktion title(). Mit title("Barplot") erstelle ich einen Titel mit dem Wortlaut Barplot, mit title("Dies ist ein Barplot\nmit Konfidenzintervallen") einen Titel, der nach dem Wort Barplot einen Zeilenumbruch hat; \n = new line.
Ich will für den Titel den vertikalen Abstand zum Panel anpassen, außerdem den Schriftschnitt (normal statt fett):
title("Barplot", line=0.5, cex.main=1.2, font.main=1)
Das Argument line ist ein spezielles Argument der Funktion title(), mit dem Sie den Titel vertikal ausrichten können. cex.main und font.main sind Grafikparameter, die ich hier als Argumente der Funktion title() verwende.
Die Einstellung cex.main=1.2 entspricht der Grundeinstellung (Schriftgröße für den Titel 20 Prozent größer als die Standardschriftgröße). Ich habe diese Angabe hier gemacht damit Sie sehen, wie Sie die Schriftgröße des Titels ändern könnten. Mögliche Schriftschnitte sind font.main gleich 1 (normal), 2 (fett), 3 (kursiv) oder 4 (fett und kursiv).
Ich erstelle jetzt die Beschriftung der X-Achse:
mtext("type", 1, line=1.3, cex=1)
Erforderliche Angaben sind die Beschriftung (hier "type") und die Seite mit 1 = unten, 2 = links, 3 = oben und 4 = rechts. Es wäre somit auch möglich, den Titel mit mtext() zu erstellen. Das Argument line steuert - wie bei der Funktion title() - den Abstand der Beschriftung vom Panel.
Der Parameter cex, der in der Grundeinstellung gleich 1 ist, steuert die Standardschriftgröße, die auch für mtext() maßgeblich ist. Die Angabe cex=1 ist hier überflüssig, sie soll zeigen, wie Sie den Schriftgrad der Achsenbeschriftung ändern könnten.
Jetzt zur Beschriftung der Y-Achse:
mtext("income", 2, line=2.3, cex=1, las=0)
Ich habe hier zusätzlich den Parameter las auf 0 gesetzt und damit festgelegt, dass der Achsentitel parallel zur Achse verlaufen soll (für die Y-Achse daher vertikal). Der Parameter las hat in der Grundeinstellung von R den Wert 0, EZR setzt den Parameter las aber standardmäßig auf 1.
Zum Abschluss will ich die Seitenränder anpassen, deren Breite durch den Parameter mar definiert ist. In der Grundeinstellung gilt mar=c(5.1,4.1,4.1,2.1). Die Seitenränder sind demnach als Vektor spezifiziert mit der ersten Komponente für den unteren Rand, der zweiten für den linken, der dritten für den oberen und der vierten für den rechten.
Ich setzte diesen Parameter für meinen Barplot mit der Funktion par() auf c(2.8,3.7,1.9,0.5), was für alle vier Seiten auf kleinere Ränder hinausläuft.
Ergebnis:
windows(width=7*390/672, height=7*390/672)
par(mar=c(2.8,3.7,1.9,0.5))
par(tcl=-0.18, cex.axis=0.8, mgp=c(3,0.2,0), fg="#666666")
barx <- barplot(bar.means, ylim=c(ifelse(min(bar.means, na.rm=TRUE)>0, 0, min(bar.means-bar.sds, na.rm=TRUE)*1.2), max(bar.means+bar.sds, na.rm=TRUE)*1.2), xlab="", ylab="", axis.lty=1, axes=FALSE, col="#ffe5cc", border="black")
par(fg="black")
error.bar(barx, bar.means, bar.sds)
axis(2, tcl=-0.2, las=1, mgp=c(3,0.4,0), col.ticks="#666666")
box(col="#666666")
title("Barplot", line=0.5, cex.main=1.2, font.main=1)
mtext("type", 1, line=1.3, cex=1)
mtext("income", 2, line=2.3, cex=1, las=0)
par(tcl=-0.5, mgp=c(3,1,0), cex.axis=1)
par(mar=c(5.1,4.1,4.1,2.1))
Ich habe die Seitenränder in der zweiten Zeile angepasst, weil diese Einstellung die High-level-Funktion barplot() betrifft, die den Bereich für das Panel anhand der Randbreiten festlegt.
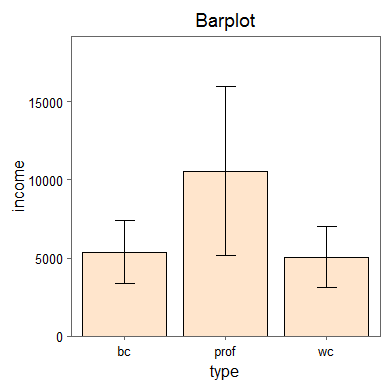
Zum Vergleich noch einmal der mit KMggplot2 erstellte Scatterplot (theme_few):

Wie Sie einzelne Grafikparameter für eine Anpassung an das theme_few setzen sollten, hängt davon ab, welches Grafikformat Sie benötigen, welche Auflösung Sie im Fall von Pixelgrafiken wünschen, was für ein Computersystem Sie nutzen und wie Sie die Grafiken erstellen (per Grafikfenster oder auf andere Weise). Eine Gebrauchsanweisung von der Art »für theme_few setzen Sie in Base Graphics die Parameter X, Y, Z auf die Werte a, b und c« wäre daher unsinnig.
Die in Base Graphics verfügbaren Grafikparameter finden Sie in der Hilfe zur Funktion par(). Tippen Sie hierzu im oberen Fenster des R Commanders (Registerkarte R Skript) die Anweisung ?par, markieren Sie diese Anweisung mit der Maus und klicken Sie auf den Button Befehl ausführen.
Sie sehen jetzt in Ihrem Standardbrowser die Hilfedatei zur Funktion par(). Interessant ist hier der Unterpunkt »Graphical Parameters«.
Im Hinblick auf den mit KMggplot2 erstellten Scatterplot sind die Erläuterungen zum Grafikparameter pch bedeutsam, weil für Base Graphics die Grundeinstellung pch=1 gilt (kleine Kreise). Für schwarze Punkte wie im Scatterplot wäre pch=16 eine gute Wahl. Beachten Sie bitte den Link in den Erläuterungen zum Grafikparameter pch.
Arbeitsschritte und Ergebnisse speichern
R-Skripte erzeugen und wiederverwenden
Sie können so gut wie alle Anweisungen, die Sie R mit dem R Commander gegeben haben in Form einer Datei mit der Endung .R speichern und bei Bedarf sehr leicht wieder einlesen und in den Arbeitsspeicher laden.
Wählen Sie im R Commander den Menüpunkt Datei / Save script oder Datei / Save script as und speichern Sie auf diese Weise den Inhalt der Registerkarte R Skript als Datei (Endung .R). Beenden Sie R und damit auch den R Commander.
Starten Sie R und den R Commander. Wählen Sie im R Commander den Menüpunkt Datei / Load skript file und öffnen Sie die gerade erstellte .R-Datei. Markieren Sie anschließend den gesamten Inhalt des Fensters R Skript (die Tasten Strg und A gleichzeitig drücken). Klicken Sie dann auf den Button Befehl ausführen.
Sie sind jetzt wieder auf dem Stand vom Ende der vorherigen Session. Über das Feld Datenmatrix (in der Menüleiste) sind alle Datensätze verfügbar, die Sie während der vorherigen Session geladen hatten. Entsprechendes gilt für das Feld Modell.
Es gibt Anweisungen, die bei einer Speicherung unberücksichtigt bleiben.
Beispiel eins:
Sie klicken auf den Button Datenmatrix bearbeiten (in der Menüleiste) und löschen mit dem Editor eine Spalte des Datensatzes Prestige. In der Registerkarte R Skript und im Fenster Ausgabe steht nur editDataset(Prestige). Aus einer .R-Datei kann daher nicht hervorgehen, was genau Sie mit dem Datensatz Prestige gemacht haben.
Eine weitere Ausnahme sind Aufforderungen, bestimmte Elemente oder Koordinaten mit der Maus zu spezifizieren.
Beispiel zwei:
Ausgangspunkt ist der Datensatz Prestige. Gehen Sie zum Menüpunkt Original menu / Grafiken / Streudiagramm … und dort zur Registerkarte Optionen. Sie haben hier in der Rubrik »Bestimmen der Punkte« die Option »Mit der Maus auswählen«. Was Sie dann im Grafikfenster anklicken bleibt in der Registerkarte R Skript unberücksichtigt.
Reports erstellen (R Markdown)
Aktivieren Sie im oberen Fenster des R Commanders die Registerkarte R Markdown. Sie sehen jetzt automatisch generierten Code, der durch Leerzeilen in Absätze untergliedert ist.
Der erste Absatz lautet standardmäßig:
---
title: "Replace with Main Title"
author: "Your Name"
date: "AUTOMATIC"
---
Alle anderen Absätze beginnen mit ```{r und enden mit ``` (drei Backticks). Diese Absätze fassen zusammen, was Sie in der aktuellen Session gemacht haben. Unberücksichtigt sind Anweisungen, die R nicht als R-Skript speichern kann (Beispiele eins und zwei im letzten Abschnitt) und Anweisungen, auf die R mit einer Fehlermeldung reagiert hat.
Klicken Sie auf den Button Erstelle Bericht. Sie sehen das Ergebnis in Ihrem Browser. Alle Anweisungen sind hellgrau hinterlegt, alle Ergebnisse entweder in separaten Boxen oder als Grafiken.
Sie können den Inhalt der Registerkarte R Markdown bearbeiten und erst danach den Bericht erstellen. Ich halte es für sinnvoll, in der Registerkarte R Markdown einen Absatz entweder zu lassen wie er ist oder komplett zu löschen. Als Ausnahme von dieser Regel sehe ich nur den ersten Absatz mit dem Titel, dem Verfassernamen und dem Datum.
Durch zusätzliche Leerzeilen vor oder nach einem Absatz schaffen Sie in der Registerkarte R Markdown Platz für zusätzlichen Text.
Überschriften erhalten Sie durch vorangestellte Raute-Zeichen:
# Überschrift 1
## Überschrift 2
Einen Zeilenumbruch erzeugen Sie durch (mindestens) zwei Leerzeichen am gewünschten Zeilenende.
Formatierung *kursiv*, Formatierung **fett**.
Verwendete Software-Versionen
Ich habe für meine Einführung in den R Commander mit folgender Software gearbeitet:
- Windows 8
- R 3.3.1 (64-bit)
- Rcmdr Version 2.2-5
- RcmdrMisc Version 1.0-4
- EZR Version 1.32
- KMggplot2 Version 0.2-3
- NMBU Version: 1.8.5
Es kann sein, dass mit anderen Software-Versionen einiges nicht so funktioniert wie in meinem Tutorial. Ich hatte zum Beispiel den Vorschlag unterbreitet, in der Datei .Rprofile die Anweisung zu geben, auf die Frage nach dem Speichern eines Workspace Images zu verzichten. Dies funktioniert mit R 3.3.1 (64-bit) für Windows einwandfrei. Anders wäre es, wenn die R-Konsole hierfür einen Menüpunkt hätte, über den Sie dies einstellen könnten.
Internetseiten
Fachbeiträge
Das liberale Propaganda-Handbuch, Taschenbuch, 382 Seiten
Einführung in die Statistik-Software R Commander
Business Cases für den Verkauf
Fachliteratur suchen mit Google Scholar, WorldCat etc. pp.
Ghostwriter für Dissertationen, Bachelor- und Masterarbeiten
Wissenschaftliches Ghostwriting
Content-Marketing mit White Papers für Start-up-Unternehmen im B2B-Geschäft
1. White Papers als Werbemittel
2. Fallstudien im Sinne von White Papers
Warum White Papers im B2B-Geschäft häufig wirkungsvoller sind als klassische Werbung
Lead-Management im B2B-Geschäft - warum und wie?
White Papers erstellen - von der Themenwahl bis zum Layout